DNA MITOCONDRIAL ¿Cual es el DNA mitocondrial?
Todas las células eucariotas poseen un organulo celular que se encarga de llevar a cabo el metabolismo oxidativo con el objetivo de la produccion de ATP, este organulo recibe el nombre de mitocondria (Lopez & Montoya, 2012). El proceso involucrado en la produccion de ATP es conocido como fosforilacion oxidativa y en el se refleja la accion de varios complejos enzimaticos (cada uno designado con un numero romano del 1 al 5) ubicados en la membrana interna de la mitocondria, gracias a ellos es posible la produccion mayoritaria del ATP celular (Fig. 1) (Lopez & Montoya, 2012).

Figura 1. Cadena transportadora de electrones en la membrana interna de la mitocondria
Una de las características mas importantes del orgánulo productor de ATP, es la existencia de una maquinaria con un sistema genético propio, en el que se sintetiza ARN, ADN y varias proteínas de codificación variable (Lopez & Montoya, 2012). Entre las proteínas que codifica se encuentran 13 correspondientes a la cadena respiratoria de la cadena proteinica de la fosforilacion oxidativa. Las mitocondrias a pesar de que tengan un sistema de genes propio codifican una cantidad mucho menor de genes que el nucleo de la celula y no son autónomas, pues tanto la formación del orgánulo como la expresion de su genoma requiere de la accion de proteinas y fragmentos de adn provenientes del núcleo. Las proteinas nucleares son sintetizadas por ribosomas en el citoplasma y son transportadas a la mitocondria (Lopez & Montoya, 2012).Existen estructuras que empaquetan varios factores de replicacion junto con hebras de ADN para su posterior transporte a la mitocondria, estas estructuras son llamadas nucleoides. Dentro de los nucleoides existe un empaquetado de cerca de 30 proteínas en varias especies animales y la parte central estaría compuesta por proteínas que participan en la síntesis de ácidos nucleicos y la parte periferica estaria compuesta de proteinas de ensamblaje. Por otro lado, todavia no esta claro el numero de moleculas de ADN que se encuentran dentro del nucleoide, pero se han realizado estudios que cuantifican de 2 a 15 moleculas de ADN dentro del mismo (Fig. 2) (Lopez & Montoya, 2012).
El ADN mitocondrial tiene una estructura duplo helicoidal circular cerrada y consta de 16000 pares de bases aproximadamente (Lopez & Montoya, 2012). Este ADN mitocondrial codifica 37 genes para ARN robosomicos, ARN de transferencia y tambien para polipeptidos quecomponen la cadena respiratoria en la mitocondria. Los RNA Ribosomicos formados pertenecen a las subunidades 12S y 16S, y se encuentra la codificacion de alrededor de 22 ARN transportadores (Lopez & Montoya, 2012). Por otro lado, existe una codificación para 13 polipeptidos, de los cuales 7 pertenecen al complejo 1 de la cadena respiratoria (designados con las abreviaturas ND1, ND2, ND3, ND4, ND4L, ND5 Y ND6), uno que corresponde al apocitocromo del complejo 3, otras 3 subunidades llamadas COX 1, COX 2 y COX 3 asociadas al complejo 4, y finalmente dos polipeptidos que corresponden a las subunidades 6 y 8 de la ATPasa (complejo 5)(Fig. 3) (Lopez & Montoya, 2012).

Figura 3. Estructura duplo helicoidal circular del ADN mitocondrial. Observese los sitios de codificacion paradistintos ARNr, ARNt y peptidos contituyentes de la cadena respiratoria
En la composicion del ADN mitocondrial es evidente la presencia de de dos cadenas, una cadena ligera, designada con la letra (L) y una cadena pesada, designada con la letra (H). La cadena pesada codifica para dos ARN ribosomicos (ARNr), 14 ARN transportadores y 12 polipeptidos, mientras que la cadena ligera codifica para 8 ARN transportadores y el polipeptido ND6 (Lopez & Montoya, 2012). Existe una zona que no codifica para ningun gen y carece de intrones, esta zona corresponde al 7% del ADN mitocondrial y es llamada bucle de desplazamiento o bucle D. El bucle D esta relacionado con el punto de origen de la replicacion y en el se encuentran los promotores de la transcripción de ambas cadenas, la cadena ligera (L) y la cadena pesada (H) (Lopez & Montoya, 2012).
PERO........¿COMO SE REPLICA EL DNA MITOCONDRIAL?
Actualmente, existen dos teorias que intentan explicar la replicacion del ADN mitondrial. La primera corresponde a un mecanismo que utiliza un modelo de desplazamiento de las cadenas, con dos origenes de replicacion; uno para la cadena pesada y otro para la cadena ligera. La segunda, obedece a un modelo con un solo origen de replicacion, que intenta ser explicado por dos variaciones llamadas: modelo acoplado y RITOLS (Lopez & Montoya, 2012). Acontinuacion se explicará cada uno de los modelos con respectivo mecanismo de funcionamiento.
1) Modelo de desplazamiento de cadenas
Es un proceso de síntesis de ADN mitocondrial unidireccional y tiene dos orígenes de replicacion, designados con las siglas (OH y OL). La sección OH se localiza en la región correspondiente el bucle de desplazamiento D, y la sección OL se encuentra a dos tercios de la longitud del punto de origen de la cadena pesada OH. La replicacion comienza con la sintesis de un ARN iniciador, el cual es formado por un ARN transcrito apartir de un promotor de la cadena ligera, este promotor se prolonga por la accion de de una proteina llamada ADN polimerasa Gamma (Lopez & Montoya, 2012). Anticipadamente, para la sisntesis de un ARN iniciador (indispensable para la transicion de ARN a ADN), fue necesaria la accion de una endoribonucleasa que corta un ARN. El ARN iniciador resultante es el sustrato para la ADN polimerasa (Fig. 4)(Lopez & Montoya, 2012)
La replicacion inicia con la síntesis de un segmento corto de una cadena H, la cual esta unida a una cadena de ADN molde. Su consecuente desplazamiento da origen a la forma de la triple hélice en el bucle D (Lopez & Montoya, 2012). La cadena H hija se elonga de forma unidireccional sobre la cadena L , al mismo tiempo que se desplaza una cadena H parental. Para la replicacion de la cadena L se necesita de una primasa especifica que solo sera activa en el punto exacto en el que la cadena H abandona el punto de origen de la replicacion OL (Fig. 5) (Lopez & Montoya, 2012).
Figura 5. Modelo de Desplazamiento por cadenas. Observese el sitio de origen de replicacion de cadena ligera (OL) y el de cadena pesada (OH). La sintesis de la cadena ligera, va despues de la sintesis de la cadena pesada. (No se observa la primasa especifica)
2) Modelo acoplado y RITOLS
El modelo acoplado plantea que existen intermediarios de replicacion de doble cadena ubicados en un unido punto de replicacion. Desde ese punto, la forma en la que se replicaban las cadenas era bidireccional (Lopez & Montoya, 2012) (Fig. 6)
Figura 9. Papeles de la ARN polimerasa monomerica y el ARNt en la mitocondria
FINALMENTE ¿COMO SE TRADUCE EL DNA MITOCONDRIAL?
Las proteinas ribosomicas estan codificadas en el genoma nuclear y se requiere de factores de traduccion como los factores de iniciacion IF2 e IF3, cuatro factores de elongacion EFTu, EFTs, EFG1 y EFG2, un factor de liberacion RF1 y un factor de reciclado ribosomal RRF (Gonzalez, 2006). Actualmente no se tiene muy claro el proceso de sintesis de proteinas en la mitocondria, sin embargo, se ha encontrado esta cantidad de factores que participan en el debido proceso para que ocurra un metabolismo optimo dentro del organulo celular.
REFERENCIAS
Gonzalez-Vioque, E., et al. (2006), 'Association of novel POLG mutations and multiple mitochondrial DNA deletions with variable clinical phenotypes in a Spanish population', Arch Neurol, 63 (1), 107-11.
Lopez, M; Montoya, J (2012) Capitulo 2. Sistema Genetico Mitocondrial Humano. Departamento de Bioquímica y Biología Molecular y Celular, Universidad de Zaragoza. Miguel Servet 177. 50013
Zaragoza., España.

Figura 1. Cadena transportadora de electrones en la membrana interna de la mitocondria
Una de las características mas importantes del orgánulo productor de ATP, es la existencia de una maquinaria con un sistema genético propio, en el que se sintetiza ARN, ADN y varias proteínas de codificación variable (Lopez & Montoya, 2012). Entre las proteínas que codifica se encuentran 13 correspondientes a la cadena respiratoria de la cadena proteinica de la fosforilacion oxidativa. Las mitocondrias a pesar de que tengan un sistema de genes propio codifican una cantidad mucho menor de genes que el nucleo de la celula y no son autónomas, pues tanto la formación del orgánulo como la expresion de su genoma requiere de la accion de proteinas y fragmentos de adn provenientes del núcleo. Las proteinas nucleares son sintetizadas por ribosomas en el citoplasma y son transportadas a la mitocondria (Lopez & Montoya, 2012).Existen estructuras que empaquetan varios factores de replicacion junto con hebras de ADN para su posterior transporte a la mitocondria, estas estructuras son llamadas nucleoides. Dentro de los nucleoides existe un empaquetado de cerca de 30 proteínas en varias especies animales y la parte central estaría compuesta por proteínas que participan en la síntesis de ácidos nucleicos y la parte periferica estaria compuesta de proteinas de ensamblaje. Por otro lado, todavia no esta claro el numero de moleculas de ADN que se encuentran dentro del nucleoide, pero se han realizado estudios que cuantifican de 2 a 15 moleculas de ADN dentro del mismo (Fig. 2) (Lopez & Montoya, 2012).
Figura 2. Composicion Quimica de un nucleoide. Observese la parte central con la presencia de proteinas implicadas en la replicacion.
El ADN mitocondrial tiene una estructura duplo helicoidal circular cerrada y consta de 16000 pares de bases aproximadamente (Lopez & Montoya, 2012). Este ADN mitocondrial codifica 37 genes para ARN robosomicos, ARN de transferencia y tambien para polipeptidos quecomponen la cadena respiratoria en la mitocondria. Los RNA Ribosomicos formados pertenecen a las subunidades 12S y 16S, y se encuentra la codificacion de alrededor de 22 ARN transportadores (Lopez & Montoya, 2012). Por otro lado, existe una codificación para 13 polipeptidos, de los cuales 7 pertenecen al complejo 1 de la cadena respiratoria (designados con las abreviaturas ND1, ND2, ND3, ND4, ND4L, ND5 Y ND6), uno que corresponde al apocitocromo del complejo 3, otras 3 subunidades llamadas COX 1, COX 2 y COX 3 asociadas al complejo 4, y finalmente dos polipeptidos que corresponden a las subunidades 6 y 8 de la ATPasa (complejo 5)(Fig. 3) (Lopez & Montoya, 2012).

Figura 3. Estructura duplo helicoidal circular del ADN mitocondrial. Observese los sitios de codificacion paradistintos ARNr, ARNt y peptidos contituyentes de la cadena respiratoria
En la composicion del ADN mitocondrial es evidente la presencia de de dos cadenas, una cadena ligera, designada con la letra (L) y una cadena pesada, designada con la letra (H). La cadena pesada codifica para dos ARN ribosomicos (ARNr), 14 ARN transportadores y 12 polipeptidos, mientras que la cadena ligera codifica para 8 ARN transportadores y el polipeptido ND6 (Lopez & Montoya, 2012). Existe una zona que no codifica para ningun gen y carece de intrones, esta zona corresponde al 7% del ADN mitocondrial y es llamada bucle de desplazamiento o bucle D. El bucle D esta relacionado con el punto de origen de la replicacion y en el se encuentran los promotores de la transcripción de ambas cadenas, la cadena ligera (L) y la cadena pesada (H) (Lopez & Montoya, 2012).
PERO........¿COMO SE REPLICA EL DNA MITOCONDRIAL?
Actualmente, existen dos teorias que intentan explicar la replicacion del ADN mitondrial. La primera corresponde a un mecanismo que utiliza un modelo de desplazamiento de las cadenas, con dos origenes de replicacion; uno para la cadena pesada y otro para la cadena ligera. La segunda, obedece a un modelo con un solo origen de replicacion, que intenta ser explicado por dos variaciones llamadas: modelo acoplado y RITOLS (Lopez & Montoya, 2012). Acontinuacion se explicará cada uno de los modelos con respectivo mecanismo de funcionamiento.
1) Modelo de desplazamiento de cadenas
Es un proceso de síntesis de ADN mitocondrial unidireccional y tiene dos orígenes de replicacion, designados con las siglas (OH y OL). La sección OH se localiza en la región correspondiente el bucle de desplazamiento D, y la sección OL se encuentra a dos tercios de la longitud del punto de origen de la cadena pesada OH. La replicacion comienza con la sintesis de un ARN iniciador, el cual es formado por un ARN transcrito apartir de un promotor de la cadena ligera, este promotor se prolonga por la accion de de una proteina llamada ADN polimerasa Gamma (Lopez & Montoya, 2012). Anticipadamente, para la sisntesis de un ARN iniciador (indispensable para la transicion de ARN a ADN), fue necesaria la accion de una endoribonucleasa que corta un ARN. El ARN iniciador resultante es el sustrato para la ADN polimerasa (Fig. 4)(Lopez & Montoya, 2012)
Figura 4. Secuencia de la replicacion por Desplazamiento de cadenas
La replicacion inicia con la síntesis de un segmento corto de una cadena H, la cual esta unida a una cadena de ADN molde. Su consecuente desplazamiento da origen a la forma de la triple hélice en el bucle D (Lopez & Montoya, 2012). La cadena H hija se elonga de forma unidireccional sobre la cadena L , al mismo tiempo que se desplaza una cadena H parental. Para la replicacion de la cadena L se necesita de una primasa especifica que solo sera activa en el punto exacto en el que la cadena H abandona el punto de origen de la replicacion OL (Fig. 5) (Lopez & Montoya, 2012).
Figura 5. Modelo de Desplazamiento por cadenas. Observese el sitio de origen de replicacion de cadena ligera (OL) y el de cadena pesada (OH). La sintesis de la cadena ligera, va despues de la sintesis de la cadena pesada. (No se observa la primasa especifica)
2) Modelo acoplado y RITOLS
El modelo acoplado plantea que existen intermediarios de replicacion de doble cadena ubicados en un unido punto de replicacion. Desde ese punto, la forma en la que se replicaban las cadenas era bidireccional (Lopez & Montoya, 2012) (Fig. 6)
Figura 6. Modelo de replicacion acoplado. Observese que la sintensis de las cadenas es bidireccional
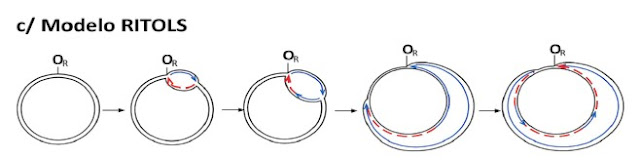
Por otro lado, el modelo RITOLS basa su fundamento gracias al descubrimiento de ribonucleotidos de cadena retrasada en fragmentos que reciben por nombre fragmentos de Okazaki, los cuales son hechas de ADN cortas recien sintetizadas. En este modelo, la replicacion se da de manera unidireccional con una cadena conductora seguida de una cadena retrasada (Fig. 7) (Lopez & Montoya, 2012).
Figura 7. Modelo de replicacion RITOLS. Observese el origen de una cadena conductora (azul) seguida de una cadena retrasada (Rojo).
La replicacion del ADN mitocondrial precisa de tres factores para s correcto funcionamiento: las proteinas de union a ADN de cadena sencilla (mtSSB), La helicasa mitocondrial y la polimerasa Gamma (Gonzalez, 2006).
1) mtSSB
Las proteinas de union de cadena sencilla tienen papel importante en la replicacacion debido a que su forma de tetramero con canales caragados positivamente permite que la cadena se ADN se estabilice al enrollarse sobre los mismos. Como consecuencia existe un aumento en la desestabilizacion de la helice por parte de la helicasa y se estimula la actividad de la ADN polimerasa (Gonzalez, 2006).
2) Helicasa mitocondrial
Su principal funcion es producir la ruptura de los enlaces de hidrogeno que comunican las dos cadenas de ADN mitocondrial, permitiendo asi la accion de la ADN polimerasa (Gonzalez, 2006)
3)PolG (Polimerasa Gamma)
La proteina mas importante en el proceso de replicacion. Posee una subunidad catalitica y una subunidad accesoria, la subunidad catalitica esta relacionada con enzimas que reparan el ADN y en ella se encuentra la actividad polimerasa y exonucleasa, o region correctora de errores (Fig. 8). Por otro lado, la subunidad accesoria aumenta la eficacia catalitica y la respectiva afinidad al ADN molde (Gonzalez, 2006).
Figura 8. Ubicacion de motivos en la subunidad catalitica en la polimerasa Gamma (motivos 1, 2 y 3 con actividad exonucleasa y motivos A, B y C con actividad polimerasa
Y LA TRANSCRIPCION DEL mtDNA ¿COMO SE DA?
Una gran diferencia entre el genoma nuclear y el genoma mitocondrial es que en el primero, la transcripcion se da gracias a tres DNA polimerasas. Por su parte, el genoma mitrocondrial requiere de la accion de la ARN polimerasa. Esta enzima genera un unico transcrito policistronico (ARN que posee posee varios codones de inicio y de parada). Adicionalmente, se crea otro transcrito menor que codifica los ARN ribosomicos correspondientes (Gonzalez, 2006). El papel de los ARN transportadores esta vinculado con el marcaje de los sitios de procesamiento de los ARN policistronicos (Fig. 9) (Gonzalez, 2006). En el humano y en los mamiferos en general se requiere de la presencia de una RNA polimerasa mitocondrial, del factor de transcripcion mitocondrial A y del factor de transcripcion mitocondrial B1 Y B2 (Gonzalez, 2006).
Adicionalmente, existe promotores de transcripcion mitocondrial que estan ubicados en sitios de control para la cadena pesada y la cadena ligera respectivamente. Estos promotores estan designados por la siglas LSP (cadena ligera) y HSP1, HSP2 (cadena pesada) (Fig. 3) (Gonzalez, 2006).
FINALMENTE ¿COMO SE TRADUCE EL DNA MITOCONDRIAL?
Las proteinas ribosomicas estan codificadas en el genoma nuclear y se requiere de factores de traduccion como los factores de iniciacion IF2 e IF3, cuatro factores de elongacion EFTu, EFTs, EFG1 y EFG2, un factor de liberacion RF1 y un factor de reciclado ribosomal RRF (Gonzalez, 2006). Actualmente no se tiene muy claro el proceso de sintesis de proteinas en la mitocondria, sin embargo, se ha encontrado esta cantidad de factores que participan en el debido proceso para que ocurra un metabolismo optimo dentro del organulo celular.
REFERENCIAS
Gonzalez-Vioque, E., et al. (2006), 'Association of novel POLG mutations and multiple mitochondrial DNA deletions with variable clinical phenotypes in a Spanish population', Arch Neurol, 63 (1), 107-11.
Lopez, M; Montoya, J (2012) Capitulo 2. Sistema Genetico Mitocondrial Humano. Departamento de Bioquímica y Biología Molecular y Celular, Universidad de Zaragoza. Miguel Servet 177. 50013
Zaragoza., España.






Comentarios
Publicar un comentario